生物催化与传感实验室
一、 研究方向
本团队现有技术人员5人,副教授4人,讲师1人,其中2人为“楚天学子”。本实验经过多年发展建立了酶基因资源发掘,分子改造和高效表达系统;构建了多种生物催化用酶的高产工程菌,实现了几种造纸、纺织、食品和饲料用酶的工业化生产;构建全细胞生物传感器用于实时、在线检测环境中的污染物如多氯联苯、二噁英等。近年来,先后承担了“863”计划、国家重点研发课题、国家自然科学基金、创新群体项目及与企业的横向项目。在Nucleic Acids Research、International Journal of Biological Macromolecules、Applied Microbiology and Biotechnology、Biotechnology for Biofuels等期刊上发表文章50篇左右。在此基础上和省内外多个企业如武汉新华扬股份有限公司、湖北安琪酵母股份有限公司等建立了紧密合作,也与中科院生物物理研究所张先恩研究员、北京化工大学张桂敏教授等团队有紧密合作,并建立了联合培养模式,每年有2-4名同学可参与联合培养。
二、 团队成员
 周玉玲,副教授, 博士生导师。2006年武汉大学博士毕业后至今在湖北大学工作。一直从事微生物与分子酶学的研究,重点聚焦蛋白和酶的基因挖掘、高效表达、益生寡糖生产、大肠杆菌分泌表达系统及毕赤酵母高效表达体系的建立。在此基础上通过基因挖掘寻找多种酶包括几丁质降解系列酶,果胶酶、纤维素酶等,以满足工业生产的多种需要;通过优化的大肠杆菌分泌表达系统,提高酶的分泌表达和单位时间内的产量,降低生产成本;通过“生物砖”等方法,构建了酶的高拷贝表达载体,提高酶在毕赤酵母中的表达。主持或参与多项国家重点研发子课题、国家自然科学基金项目及省市科技项目,并获得过两项省部级科技奖励,发表文章20多篇。Email:zyl609@hubu.edu.cn
周玉玲,副教授, 博士生导师。2006年武汉大学博士毕业后至今在湖北大学工作。一直从事微生物与分子酶学的研究,重点聚焦蛋白和酶的基因挖掘、高效表达、益生寡糖生产、大肠杆菌分泌表达系统及毕赤酵母高效表达体系的建立。在此基础上通过基因挖掘寻找多种酶包括几丁质降解系列酶,果胶酶、纤维素酶等,以满足工业生产的多种需要;通过优化的大肠杆菌分泌表达系统,提高酶的分泌表达和单位时间内的产量,降低生产成本;通过“生物砖”等方法,构建了酶的高拷贝表达载体,提高酶在毕赤酵母中的表达。主持或参与多项国家重点研发子课题、国家自然科学基金项目及省市科技项目,并获得过两项省部级科技奖励,发表文章20多篇。Email:zyl609@hubu.edu.cn
 蒋思婧,副教授,硕士生导师。1989年,华东师范大学生物学本科毕业,获学士学位,1999年湖北大学硕士毕业, 2010年华农农业大学博士毕业;自1999进入湖北大学生命科学学院工作以来,一直从事微生物基因工程与分子酶学的研究,重点聚焦蛋白质和酶基因资源的挖掘和高效表达、大肠杆菌分泌表达系统及毕赤酵母高效表达体系的建立、益生因子和疫苗的开发利用等:通过基因挖掘多种适宜工业生产的酶,如系列几丁质降解酶,纤维素酶和半纤维素酶等;通过优化的大肠杆菌分泌表达系统,提高酶的分泌表达和单位时间内的产量,降低生产成本;通过“生物砖”等方法,构建了酶的高拷贝表达载体,提高酶在毕赤酵母中的表达。主持或参与多项国家重点研发子课题、国家自然科学基金项目及省市科技项目,并获得过两项省部级科技奖励,发表文章10余篇。Email:jiangsijign@hubu.edu.cn
蒋思婧,副教授,硕士生导师。1989年,华东师范大学生物学本科毕业,获学士学位,1999年湖北大学硕士毕业, 2010年华农农业大学博士毕业;自1999进入湖北大学生命科学学院工作以来,一直从事微生物基因工程与分子酶学的研究,重点聚焦蛋白质和酶基因资源的挖掘和高效表达、大肠杆菌分泌表达系统及毕赤酵母高效表达体系的建立、益生因子和疫苗的开发利用等:通过基因挖掘多种适宜工业生产的酶,如系列几丁质降解酶,纤维素酶和半纤维素酶等;通过优化的大肠杆菌分泌表达系统,提高酶的分泌表达和单位时间内的产量,降低生产成本;通过“生物砖”等方法,构建了酶的高拷贝表达载体,提高酶在毕赤酵母中的表达。主持或参与多项国家重点研发子课题、国家自然科学基金项目及省市科技项目,并获得过两项省部级科技奖励,发表文章10余篇。Email:jiangsijign@hubu.edu.cn

贺妮莎,副教授,硕士生导师,省级人才获得者。2008.09-2012.06 湖北大学生命科学学院,生物学学士;2012.09-2018.01 中国科学院上海植物生理生态研究所,微生物学博士(导师:肖友利研究员);2018.03 -2020.03 College de France,博士后(合作导师:Marc Fontecave院士);2019.03至今 湖北大学生命科学学院,副教授。主要致力于天然产物生物合成和酶催化机制、酶活性改造等方面的研究。目前主持国家自然科学基金1项,湖北省级人才项目1项,湖北省教育厅青年项目1项,在国际知名期刊Chemical Science、Nucleic Acid Research、PNAS等期刊发表学术论文8篇。Email: nshe1992@163.com
 卢争辉,副教授,硕士生导师,楚天学子,武汉青年英才。2008.09- 2012.06 湖北大学生科院,生物工程学士;2012.09-2015.06 湖北大学生科院,微生物学硕士,导师张桂敏教授;2015.09-2018.06 湖北大学生科院,获微生物学博士学位,导师张桂敏教授;2018.07-2019.07 湖北大学生科院,博士后,合作导师易犁教授;2019.07-至今,湖北大学生科院。主要致力于枯草芽孢杆菌表达系统与遗传体系、酶活性改造等方面的研究。目前主持国家自然科学基金1项,中国博士后科学基金1项,湖北省自然科学基金1项,在国际知名期刊Nucleic Acid Research、Biotechnology for Biofuels等期刊发表学术论文十余篇。Email:mailto:zhenghuilu@hubu.edu.cn
卢争辉,副教授,硕士生导师,楚天学子,武汉青年英才。2008.09- 2012.06 湖北大学生科院,生物工程学士;2012.09-2015.06 湖北大学生科院,微生物学硕士,导师张桂敏教授;2015.09-2018.06 湖北大学生科院,获微生物学博士学位,导师张桂敏教授;2018.07-2019.07 湖北大学生科院,博士后,合作导师易犁教授;2019.07-至今,湖北大学生科院。主要致力于枯草芽孢杆菌表达系统与遗传体系、酶活性改造等方面的研究。目前主持国家自然科学基金1项,中国博士后科学基金1项,湖北省自然科学基金1项,在国际知名期刊Nucleic Acid Research、Biotechnology for Biofuels等期刊发表学术论文十余篇。Email:mailto:zhenghuilu@hubu.edu.cn

巫攀,讲师,硕士生导师。2017年武汉大学博士毕业后至今在湖北大学工作。主要从事真菌毒素和持久性有毒污染物的生物降解,工业酶的基因挖掘、高效表达和分子改造。主持或参与多项科技部重点研发课题,国家自然科学基金项目及省市项目,发表文章十余篇。Email: wupan@hubu.edu.cn
三、 研究成果
1) 生物造纸用酶
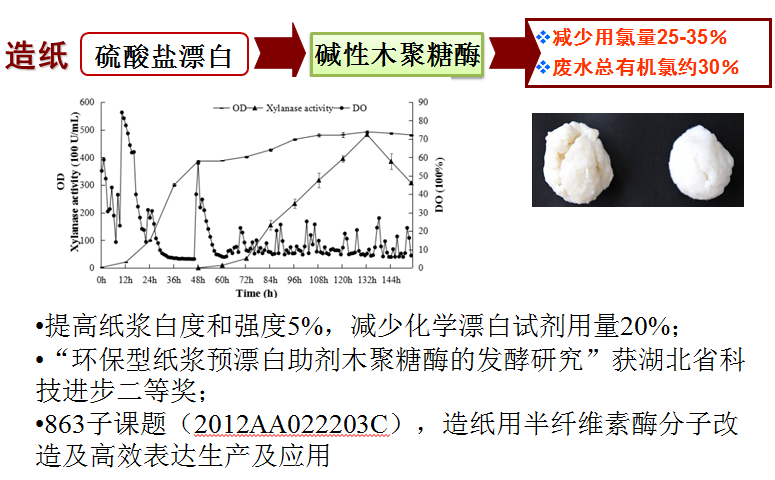
2) 纺织精炼用酶
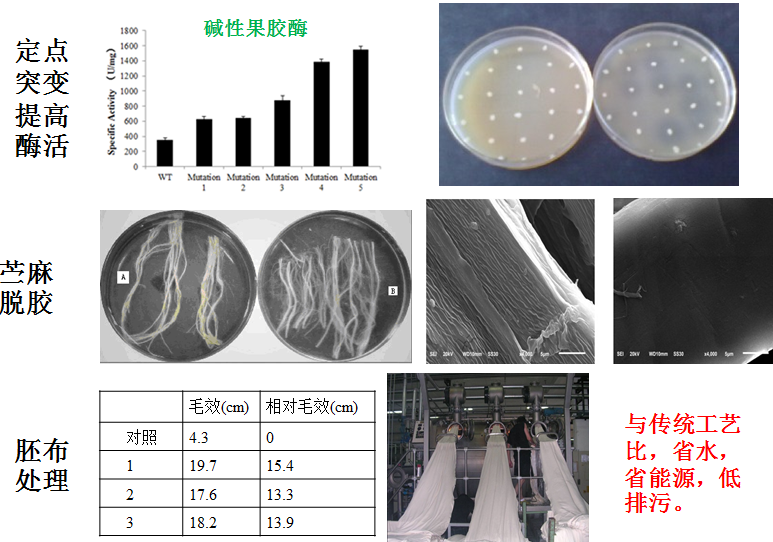
3) 酶法制备几丁寡糖系列
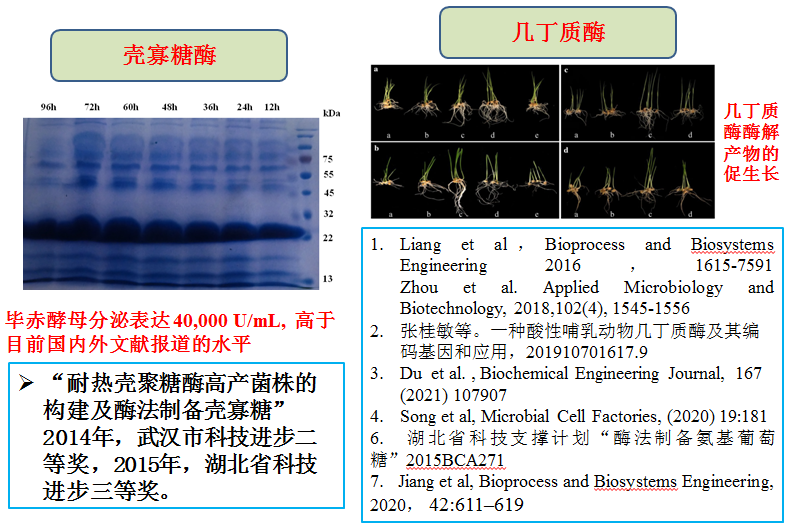
4) 真菌毒素玉米赤霉烯酮降解
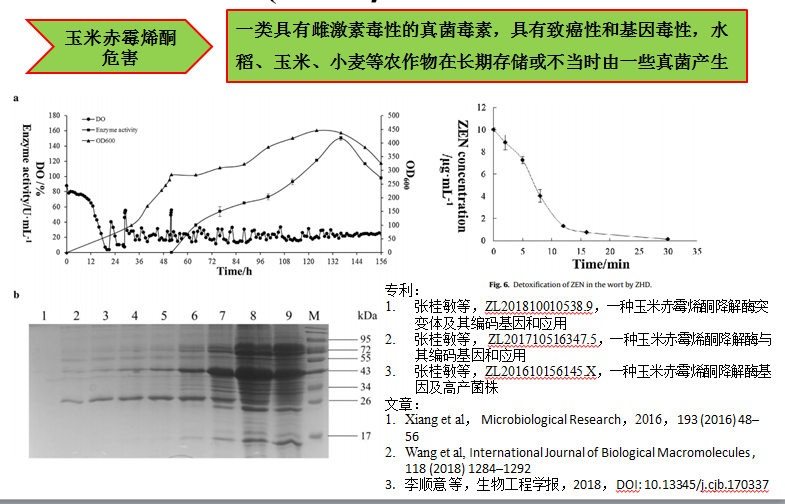
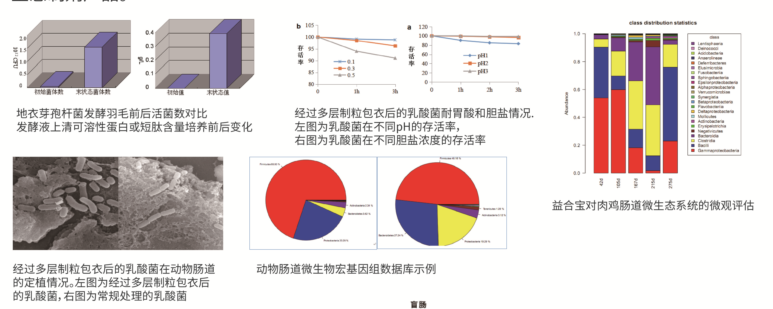
5) 废弃物发酵制备饲用微生态制剂关键技术
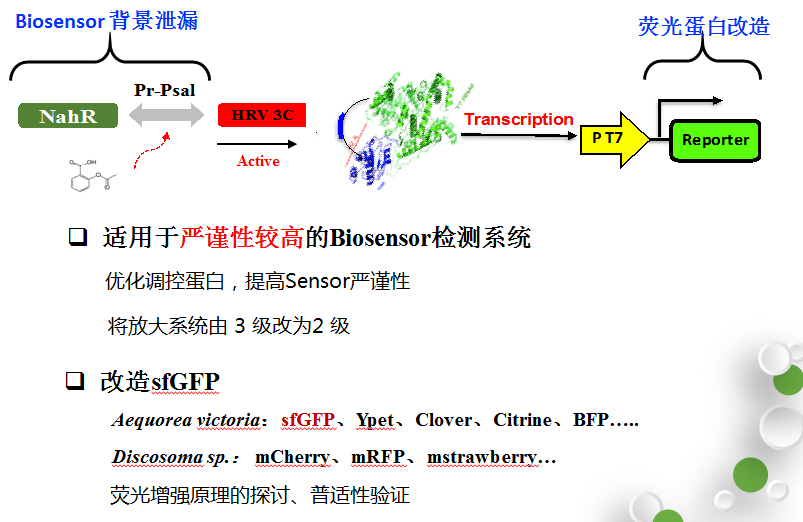
6) 全细胞生物传感构建
四、 承担的科研项目
1. 国家自然科学基金青年项目(31901031,2020-2022):感受态形成促进串联基因复制的分子机理。
2. 中国博士后科学基金面上项目(2019M662571,2019-2020):转录因子ComK 调控枯草杆菌胁迫压力应答的分子机理研究。
3. 湖北省自然科学基金面上项目(青年)(2019CFB176,2019-2020):H100Q 引起碱性木聚糖酶 Xyn10A 碱耐受性改变的机制研究。
4. 国家自然科学基金委员会与英国皇家学会合作交流项目(IEC\NSFC\181305,2020.01-2022.12):通过小角 X 射线散射和中子散射(SAXS-SANS)研究N-端结构域影响淀粉酶 Amy703 钙离子依赖性的机制。
5. 国家自然科学基金(31240008,2012.01-2012.12):糖基化对烟曲壳聚糖酶性质影响的结构生物学研究。
6. 武汉市科技攻关计划(201120722216-2):饲料抗菌肽添加剂的研究和开发。
7. 国家重点研发计划子课题 (2019YFA0905000,2020 年 01 月 - 2024 年 12 月):新分子生化反应设计与生物合成系统创建。
8. 国家自然科学基金面上项目(31670069,2017/01-2020/12),嗜碱芽孢杆菌GH13新亚家族碱性淀粉酶N端结构域的功能和进化机制研究。
9. 湖北省教育厅中青年项目(202010701301003,2020.01–2021.12),多氧霉素生物合成途径中关键成环机制的研究。
10. 国家自然科学基金面上项目(21572243,2016.01–2019.12),碳磷裂解酶参与的碳磷键断裂自由基反应机制研究。
11. 湖北省科技厅支撑计划(2015BCA271,2015/12-2017/12),酶法制备氨基葡萄糖。
12. 863子课题(2014.01-2016.12),蛋白质高拷贝表达与芽孢杆菌表达系统的构建与应用。
13. 国家自然科学基金面上项目(21572243,2016.01–2019.12),碳磷裂解酶参与的碳磷键断裂自由基反应机制研究。
14. 国家自然科学基金主任基金(21342013,2014.01–2014.12),PhnJ催化的有机膦化合物中碳磷键断裂的酶反应机制研究。
15. 国家“863”计划项目“CO2-油藻-生物柴油关键技术研究”子课题(2009AA064401,2010–2011):产油微藻的RNAi载体的规模化的构建及相关基因功能的研究。
16. 湖北省教育厅重点项目(D200710004,2007–2009):系列β-甘露聚糖酶基因的克隆和表达研究。
17. 武汉市科技攻关项目(20042001020,2004–2005):饲料酶添加剂-植酸酶的中试研究。
18. 湖北省自然科学基金(2003ABA118):烟草和水稻的质体载体的构建及质体转烟草的研究。
19. 湖北省科技攻关计划(2003AA101C22,2003–2005):应用基因工程技术生产果寡糖中试。
五、 论文
1. 周玉玲,蒋思婧,贺妮莎,张桂敏,微生物几丁质酶研究进展及其在N-乙酰氨基葡萄糖制备中的应用, 微生物学报, 2021, 61(8): 2192-2204 (期刊论文)
2. Li C, Jiang S, Du C, Lu Z, He N, Zhou Y, Zhang G*. (2021) High-level extracellular expression of a new β-N-Acetylglucosaminidase in Escherichia coli for producing GlcNAc. Frontiers in Microbiology 12, 478.
3. Chao Du,Xiang Zhao,Wen Song,Nisha He,Sijin Jiang,Yuling Zhou*, Guimin Zhang,Combined strategies to improve the expression of acidic mammalian chitinase in Pichia pastoris for the production of N, N-diacetylchitobiose, Biochemical Engineering Journal, 2021, 167(107907)
4. Song, W, Zhang, N, Yang, M, Zhou, Y, He N*, Zhang G*. (2020) Multiple strategies to improve the yield of chitinase a from Bacillus licheniformis in Pichia pastoris to obtain plant growth enhancer and GlcNAc. Microbial Cell Factories 19, 1–11.
5. Cheng Y, Xu J, Fu Y, He N*. (2020) Expression and regulation of pde6h by thyroid hormone during metamorphosis in Paralichthys olivaceus. Frontiers in Physiology 11, 244.
6. Wu P, Luo F, Lu Z, Zhan Z, Zhang G*. (2020) Improving the catalytic performance of pectate lyase through pectate lyase/Cu3(PO4)2 hybrid nanoflowers as an immobilized enzyme. Frontiers in Bioengineering and Biotechnology 8, 280.
7. Hu X, Yuan X, He N, Zhuang TZ, Wu P, Zhan, G*. (2019) Expression of Bacillus licheniformis α–amylase in Pichia pastoris without antibiotics-resistant gene and effects of glycosylation on the enzymic thermostability. 3 Biotech 9, 1–9.
8. Chao Du, Shun Jiang, Sijing Jiang,Yuling Zhou*,Guimin Zhang , A Bacillus pumilus originated β-N-acetylglucosaminidase for chitin combinatory hydrolysis and exploration of its thermosta ble mechanism, International Journal of Biological Macromolecules, 2019, 132: 1282-1289
9. Jiang S, Jiang H, Zhou Y, Jiang S*, Zhang G*. (2019). High-level expression of β-N-Acetylglucosaminidase BsNagZ in Pichia pastoris to obtain GlcNAc. Bioprocess and Biosystems Engineering 42, 611–619.
10. Lu Z, Yang S, Yuan X, Shi Y, Ouyang L, Jiang S, Yi L, Zhang G*. (2019) CRISPR-assisted multi-dimensional regulation for fine-tuning gene expression in Bacillus subtilis. Nucleic acids research 47, e40–e40.
11. Lu Z, Li X, Zhang R, Yi L, Ma Y, Zhang G*. (2019) Tunnel engineering to accelerate product release for better biomass-degrading abilities in lignocellulolytic enzymes. Biotechnology for Biofuels 12, 1–9.
12. Lu Z, Hu X, Shen P, Wang Q, Zhou Y, Zhang G*, Ma Y. (2018) A pH-stable, detergent and chelator resistant type I pullulanase from Bacillus pseudofirmus 703 with high catalytic efficiency. International Journal of Biological Macromolecules 109, 1302–1310.
13. Wang X, Lu Z, Xu T, Selvaraj JN, Yi L, Zhang G*. (2018) Improving the specific activity and thermo-stability of alkaline pectatelyase from Bacillus subtilis 168 for bioscouring. Biochemical Engineering Journal 129, 74–83.
14. Yuling Zhou, Zhenghui Lu,Xiang Wang,Jonathan Nimal Selvaraj, Guimin Zhang,Genetic engineering modification and fermentation optimization for extracellular production of recombinant proteins using Escherichia coli, Applied Microbiology and Biotechnology, 2017, 102(4): 1545-1556
15. He N#, Wu P#, Lei Y, Xu B, Zhu X, Xu G, Chen W*, Xiao Y*. (2017) Construction of an octosyl acid backbone catalyzed by a radical S-adenosylmethionine enzyme and a phosphatase in the biosynthesis of high–carbon sugar nucleoside antibiotics. Chemical science 8, 444–451.
16. Huang Liang,Wang Qinhong,Jiang Sijing,Yuling Zhou*,Zhang Guimin, Ma Yanhe,Improved extracellular expression and high-cell-density fed-batch fermentation of chitosanase from Aspergillus Fumigatus in Escherichia coli, Bioprocess and Biosystems Engineering, 2016, 39(11): 1679-168
17. Qi X, Lin W, Ma M, Wang C, He Y, He N, Zhang P* (2016) Structural basis of rifampin inactivation by rifampin phosphotransferase. Proceedings of the National Academy of Sciences of the United States of America 113, 3803–3808.
18. Lu Z, Wang Q, Jiang S, Zhang G*, Ma Y. (2016) Truncation of the unique N-terminal domain improved the thermos-stability and specific activity of alkaline α-amylase Amy703. Scientific Reports 6, 22465.
19. Zhan X, Wang Q, Jiang S, Zhou Y, Zhang G, Ma Y. (2014) Developing a xylanase XYNZG from Plectosphaerella cucumerina for baking by heterologously expressed in Kluyveromyces lactis. BMC Biotechnology 14, 1–9.
20. Lu Z, Tian C, Li A, Zhang G*, Ma Y. (2014) Identification and characterization of a novel alkaline α-amylase Amy703 belonging to a new clade from Bacillus pseudofirmus. Journal of Industrial Microbiology & Biotechnology 41, 783–793.
六、 专利
1. 张桂敏,卢争辉,沈盼盼,周玉玲,马延和。针对淀粉酶 BLA 的启动子文库及强启动子。专利授权号:ZL201710956947.3。
2. 张桂敏,卢争辉,袁芯,彭文舫,石云云,蒋思婧,马延和。一种特异性调节枯草芽孢杆菌外源基因表达的 dcas9-ω 融合蛋白及其应用。专利授权号:201710965231.X3。
3. 张桂敏,卢争辉,周玉玲,马延和。一种启动子文库构建方法和强启动子。专利授权号:ZL2015107992498 。
4. 张桂敏,卢争辉,马延和。一种热稳定性及比酶活提高的碱性淀粉酶突变体。专利授权号:ZL201410852880.5 。
5. 蔡得田,陈冬玲,宋兆建,高青青,贺妮莎,卢争辉,徐向君。利用远缘杂交和回交技术选育鸡冠花品种的方法。专利授权号:ZL 201110078292.7。
6. 张桂敏,汪声晨,蒋思婧,马延和. 一种外切-α-1,4-糖苷酶与其编码基因和应用,201610146913。
7. 张桂敏,唐雨蒙,易犁,蒋思婧,马延和. 一种低温碱性果胶裂解酶及其编码基因和应用,ZL201710497480.0。
8. 张桂敏,胡鑫霖,毛良伟,蒋思婧,马延和. 一种淀粉酶在高盐浓度条件下水解淀粉的应用和方法,201910456638.9。
9. 张桂敏,王辉,蒋思婧,王美星,马延和. 玉米赤霉烯酮降解酶在水解玉米赤霉烯酮及其衍生物中的应用,201910938276.7。
10. 马立新,蒋思婧. 一种酵母基因工程菌及内切菊粉酶制剂和应用方法,2004.10.13,中国,ZL01128440.4。
11. 马立新,蒋思婧. 一种零背景高通量定向表达克隆的方法,2004.09.08,中国,ZL01133518.1。
12. 马立新,蒋思婧. 一种基因克隆的高效方法, 2005.02.09,中国,ZL01133517.3。




